宏基因组学又叫微生物环境基因组学、元基因组学,主要基于无需预先培养来利用环境样品基因组资源,对特定环境中全部微生物的总脱氧核糖核酸(Deoxyribonucleic acid, DNA)(也称宏基因组,metagenomic)进行克隆,并通过构建宏基因组文库和筛选等手段获得新的生理活性物质和功能基因。BIOTREE开发的“四界微生态”组学,在宏基因组学的基础上进行深度微生物组学研究,同时涵盖宏细菌组、宏古菌组、宏病毒组和宏真菌组共四种单独的组学产品。为助力老师更好的深入研究肠道菌群,“世界肠道健康日”来临之际特推出特价优惠活动。

图1 活动海报
采用Illumina 测序平台、PE150 模式进行测序,测序得到的下机数据(Raw Data)将用于后期信息分析。

图2 实验处理流程
测序数据下机经质控去除测序接头、低质量序列及宿主污染后,分别进行宏细菌组、宏古菌组、宏真菌组和宏病毒组的分析。
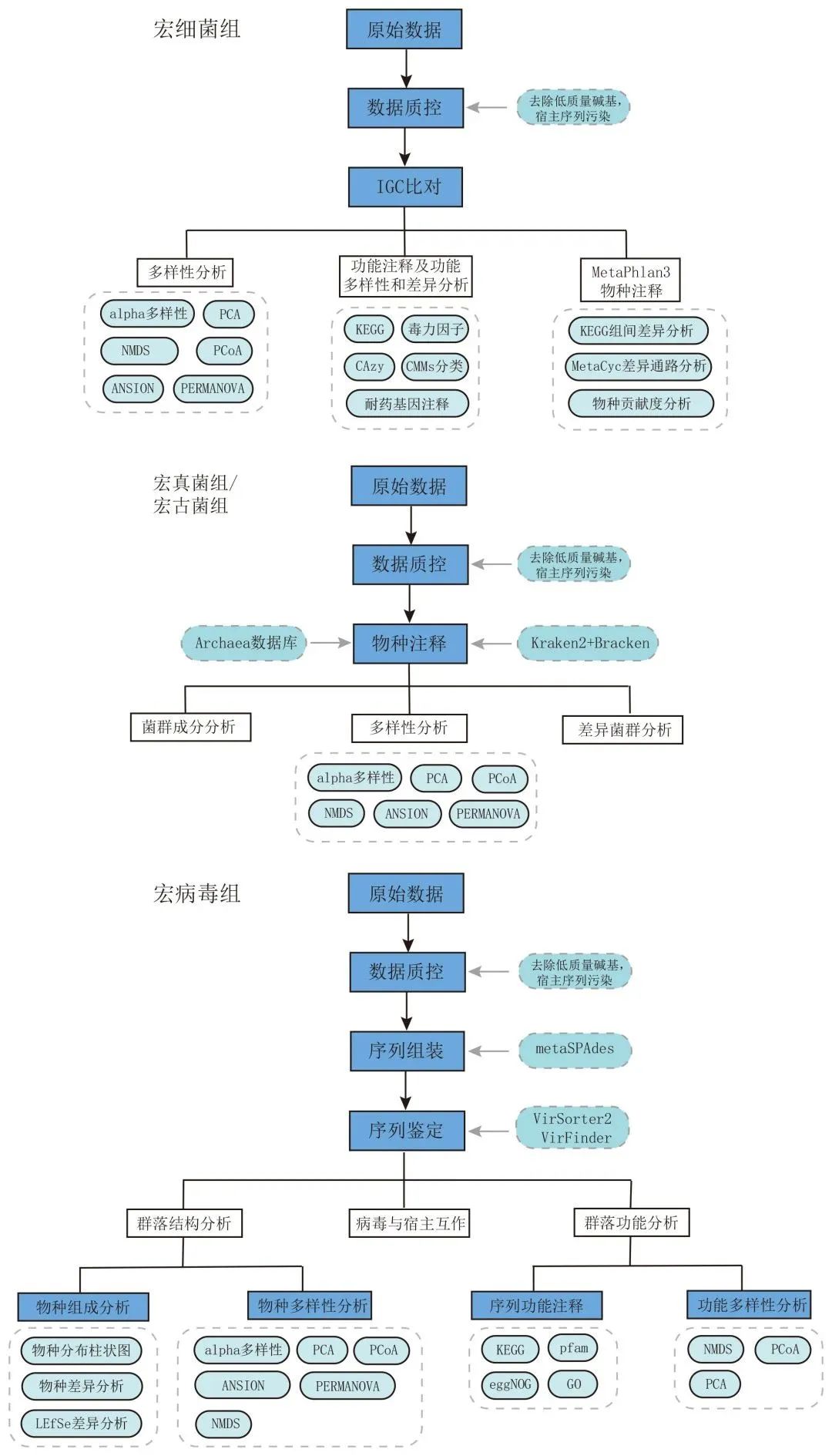
图3 分析流程
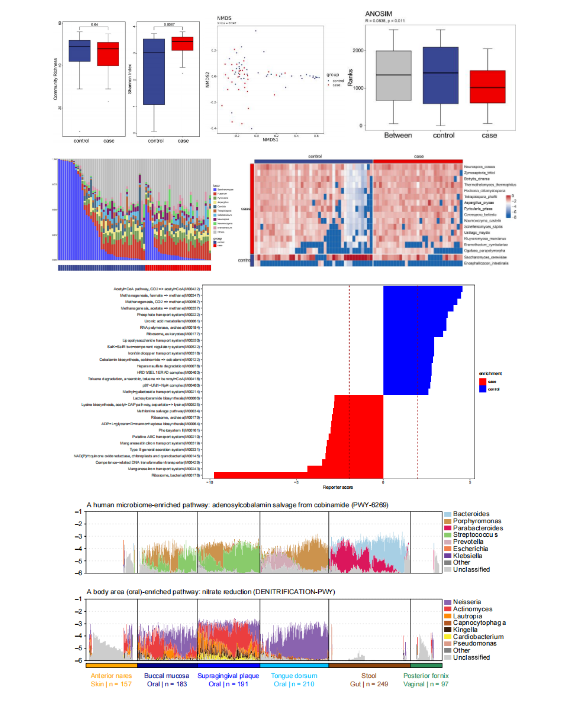
图4 部分结果图展示
文章标题:Multi-kingdom microbiota analyses identify bacterial–fungal interactions and biomarkers of colorectal cancer across cohorts
中文标题:多界微生物分析鉴别不同队列结直肠癌的细菌-真菌相互作用和生物标志物研究
文章简介:该研究通过对多中心队列的结直肠癌(colorectal cancer, CRC)患者的粪便样本进行宏基因组分析,绘制了8个结直肠癌人群队列(中国、法国、德国、奥地利、意大利、美国和日本)的1368个样本(除中国之外的人群为网站获取公开数据)的4界微生态(细菌、真菌、古菌、病毒)图谱,揭示了与CRC相关的“四界肠道微生物”和功能的变化及彼此的可能互作机制,并建立了基于多界微生物的CRC诊断模型;最后也对肠道菌群介导的功能进行研究并通过qPCR对相关基因进行验证。为结直肠癌早期诊断和预后评估及潜在的治疗靶点提供了基于微生物及其功能标志物的新方法和新思路。
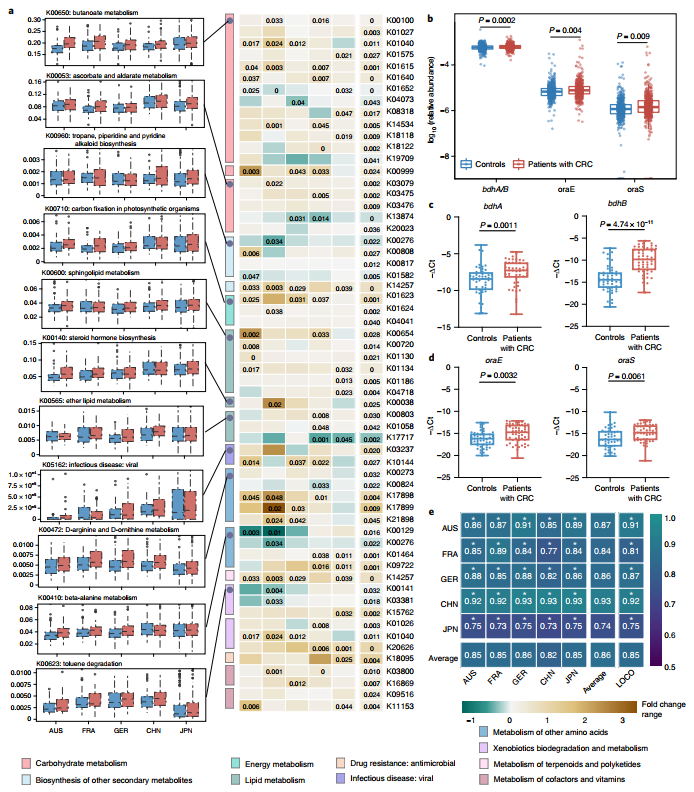
图5 部分分析结果
参考文献:
[2].Charles Y Chiu, Steven A Miller. Clinical metagenomics. Nat Rev Genet . 2019 Jun;20(6):341-355.